[TOC]
* 在数据处理中,经常会遇到特征维度比样本数量多得多的情况,如果拿到实际工程中去跑,效果不一定好。一是因为冗余的特征会带来一些噪音,影响计算的结果;二是因为无关的特征会加大计算量,耗费时间和资源。所以我们通常会对数据重新变换一下,再跑模型。**数据变换的目的不仅仅是降维,还可以消除特征之间的相关性,并发现一些潜在的特征变量。**
## 一、PCA的目的
* PCA是一种在尽可能减少信息损失的情况下找到某种方式降低数据的维度的方法。通常来说,我们期望得到的结果,是把原始数据的特征空间(n个d维样本)投影到一个小一点的子空间里去,并尽可能表达的很好(就是说损失信息最少)。常见的应用在于模式识别中,我们可以通过减少特征空间的维度,抽取子空间的数据来最好的表达我们的数据,从而减少参数估计的误差。注意,主成分分析通常会得到协方差矩阵和相关矩阵。这些矩阵可以通过原始数据计算出来。协方差矩阵包含平方和与向量积的和。相关矩阵与协方差矩阵类似,但是第一个变量,也就是第一列,是标准化后的数据。**如果变量之间的方差很大,或者变量的量纲不统一,我们必须先标准化再进行主成分分析。**
## 二、PCA VS MDA
* 提到PCA,可能有些人会想到MDA(Multiple Discriminate Analysis,多元判别分析法),这两者都是线性变换,而且很相似。只不过在PCA中,我们是找到一个成分(方向)来把我们的数据最大化方差,而在MDA中,我们的目标是最大化不同类别之间的差异(比如说,在模式识别问题中,我们的数据包含多个类别,与两个主成分的PCA相比,这就忽略了类别标签)。
* 换句话说,通过PCA,我们把整个数据集(不含类别标签)投射到一个不同的子空间中,在MDA中,我们试图决定一个合适的子空间来区分不同类别。再换种方式说,PCA是找到数据传播最广的时候的最大方差的轴axis,MDA是最大化类别与类别之间的区别。
* 上文我们提到了子空间,那么怎么样去寻找“好的”子空间呢?
* 假设我们的目标是减少d维的数据集,将其投影到k维的子空间上(看k<d)。所以,我们如何来确定k呢?如何知道我们选择的特征空间能够很好的表达原始数据呢?
* 下文中我们会计算数据中的特征向量(主成分),然后计算散布矩阵([scatter\_matrix](https://en.wikipedia.org/wiki/Scatter_matrix))中(也可以从协方差矩阵中计算)。每个特征向量与特征值相关,即特征向量的“长度”或“大小”。如果发现每个特征值都很小,那就可以说明我们的原始数据就已经是一个“好的”空间了。但是,如果有些特征值比其他值要大得多,我们只需要关注那些特别大的特征值,因为这些值包含了数据分布情况的绝大部分信息。反之,那些接近于0的特征值包含的信息几乎没有,在新的特征空间里我们可以忽略不计。
## 三、PCA的过程
* 通常来说有以下六步:
> 1.去掉数据的类别特征(label),将去掉后的d维数据作为样本
2.计算d维的均值向量(即所有数据的每一维向量的均值)
3.计算所有数据的散布矩阵(或者协方差矩阵)
4.计算特征值(e1,e2,...,ed)以及相应的特征向量(lambda1,lambda2,...,lambda d)
5.按照特征值的大小对特征向量降序排序,选择前k个最大的特征向量,组成d\*k维的矩阵W(其中每一列代表一个特征向量)
6.运用d\*K的特征向量矩阵W将样本数据变换成新的子空间。(用数学式子表达就是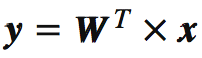,其中x是d\*1维的向量,代表一个样本,y是K\*1维的在新的子空间里的向量)
### 1.数据准备----生成三维样本向量
* 首先随机生成40\*3维的数据,符合多元高斯分布。假设数据被分为两类,其中一半类别为w1,另一半类别为w2
```
#coding:utf-8
import numpy as np
np.random.seed(4294967295)
mu_vec1 = np.array([0,0,0])
cov_mat1 = np.array([[1,0,0],[0,1,0],[0,0,1]])
class1_sample = np.random.multivariate_normal(mu_vec1, cov_mat1, 20).T
assert class1_sample.shape == (3,20)#检验数据的维度是否为3*20,若不为3*20,则抛出异常
mu_vec2 = np.array([1,1,1])
cov_mat2 = np.array([[1,0,0],[0,1,0],[0,0,1]])
class2_sample = np.random.multivariate_normal(mu_vec2, cov_mat2, 20).T
assert class1_sample.shape == (3,20)#检验数据的维度是否为3*20,若不为3*20,则抛出异常
```
* 运行这段代码后,我们就生成了包含两个类别的样本数据,其中每一列都是一个三维的向量,所有数据是这样的矩阵:
* 结果:
### 2.作图查看原始数据的分布
```
from matplotlib import pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from mpl_toolkits.mplot3d import proj3d
fig = plt.figure(figsize=(8,8))
ax = fig.add_subplot(111, projection='3d')
plt.rcParams['legend.fontsize'] = 10
ax.plot(class1_sample[0,:], class1_sample[1,:], class1_sample[2,:], 'o', markersize=8, color='blue', alpha=0.5, label='class1')
ax.plot(class2_sample[0,:], class2_sample[1,:], class2_sample[2,:], '^', markersize=8, alpha=0.5, color='red', label='class2')
plt.title('Samples for class 1 and class 2')
ax.legend(loc='upper right')
plt.show()
```
* 结果:
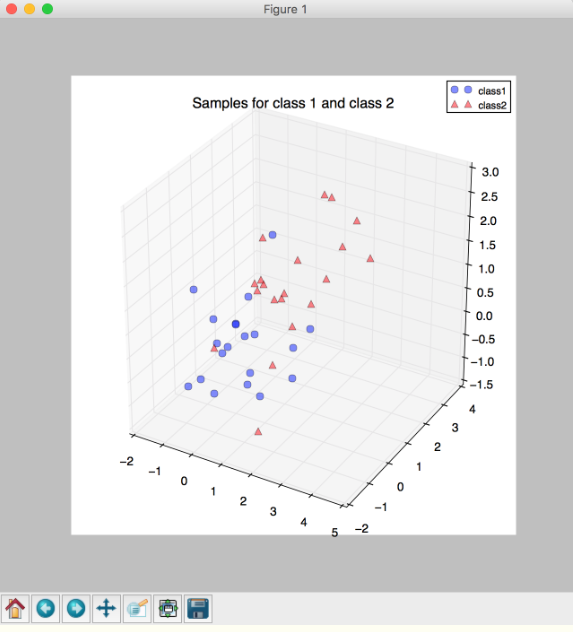
### 3.去掉数据的类别特征
```
1 all_samples = np.concatenate((class1_sample, class2_sample), axis=1)
2 assert all_samples.shape == (3,40)#检验数据的维度是否为3*20,若不为3*20,则抛出异常
```
### 4.计算d维向量均值
```
mean_x = np.mean(all_samples[0,:])
mean_y = np.mean(all_samples[1,:])
mean_z = np.mean(all_samples[2,:])
mean_vector = np.array([[mean_x],[mean_y],[mean_z]])
print('Mean Vector:\n', mean_vector)
```
* 结果:
```
1 print('Mean Vector:\n', mean_vector)
2 Mean Vector:,
3 array([[ 0.68047077],
4 [ 0.52975093],
5 [ 0.43787182]]))
```
### 5.计算散步矩阵或者协方差矩阵
* a.计算散步矩阵
* 散布矩阵公式:
* 其中m是向量的均值:(第4步已经算出来是mean\_vector)
```
1 scatter_matrix = np.zeros((3,3))
2 for i in range(all_samples.shape[1]):
3 scatter_matrix += (all_samples[:,i].reshape(3,1) - mean_vector).dot((all_samples[:,i].reshape(3,1) - mean_vector).T)
4 print('Scatter Matrix:\n', scatter_matrix)
```
* 结果:
```
1 print('Scatter Matrix:\n', scatter_matrix)
2 ('Scatter Matrix:,
3 array([[ 46.81069724, 13.95578062, 27.08660175],
4 [ 13.95578062, 48.28401947, 11.32856266],
5 [ 27.08660175, 11.32856266, 50.51724488]]))
```
* b.计算协方差矩阵
* 如果不计算散布矩阵的话,也可以用python里内置的numpy.cov()函数直接计算协方差矩阵。因为散步矩阵和协方差矩阵非常类似,散布矩阵乘以(1/N-1)就是协方差,所以他们的特征空间是完全等价的(特征向量相同,特征值用一个常数(1/N-1,这里是1/39)等价缩放了)。协方差矩阵如下所示:
```
1 cov_mat = np.cov([all_samples[0,:],all_samples[1,:],all_samples[2,:]])
2 print('Covariance Matrix:\n', cov_mat)
```
* 结果:
```
1 >>> print('Covariance Matrix:\n', cov_mat)
2 Covariance Matrix:,
3 array([[ 1.20027429, 0.35784053, 0.69452825],
4 [ 0.35784053, 1.23805178, 0.29047597],
5 [ 0.69452825, 0.29047597, 1.29531397]]))
```
### 6.计算相应的特征向量和特征值
```
1 # 通过散布矩阵计算特征值和特征向量
2 eig_val_sc, eig_vec_sc = np.linalg.eig(scatter_matrix)
3
4 # 通过协方差矩阵计算特征值和特征向量
5 eig_val_cov, eig_vec_cov = np.linalg.eig(cov_mat)
6
7 for i in range(len(eig_val_sc)):
8 eigvec_sc = eig_vec_sc[:,i].reshape(1,3).T
9 eigvec_cov = eig_vec_cov[:,i].reshape(1,3).T
10 assert eigvec_sc.all() == eigvec_cov.all()
11
12 print('Eigenvector {}: \n{}'.format(i+1, eigvec_sc))
13 print('Eigenvalue {} from scatter matrix: {}'.format(i+1, eig_val_sc[i]))
14 print('Eigenvalue {} from covariance matrix: {}'.format(i+1, eig_val_cov[i]))
15 print('Scaling factor: ', eig_val_sc[i]/eig_val_cov[i])
16 print(40 * '-')
```
* 结果:
```
1 Eigenvector 1:
2 [[-0.84190486]
3 [-0.39978877]
4 [-0.36244329]]
5 Eigenvalue 1 from scatter matrix: 55.398855957302445
6 Eigenvalue 1 from covariance matrix: 1.4204834860846791
7 Scaling factor: 39.0
8 ----------------------------------------
9 Eigenvector 2:
10 [[-0.44565232]
11 [ 0.13637858]
12 [ 0.88475697]]
13 Eigenvalue 2 from scatter matrix: 32.42754801292286
14 Eigenvalue 2 from covariance matrix: 0.8314755900749456
15 Scaling factor: 39.0
16 ----------------------------------------
17 Eigenvector 3:
18 [[ 0.30428639]
19 [-0.90640489]
20 [ 0.29298458]]
21 Eigenvalue 3 from scatter matrix: 34.65493432806495
22 Eigenvalue 3 from covariance matrix: 0.8885880596939733
23 Scaling factor: 39.0
24 ----------------------------------------
```
* 其实从上面的结果就可以发现,通过散布矩阵和协方差矩阵计算的特征空间相同,协方差矩阵的特征值\*39 = 散布矩阵的特征值。
* 当然,我们也可以快速验证一下特征值-特征向量的计算是否正确,是不是满足方程(其中为协方差矩阵,v为特征向量,lambda为特征值)。
```
1 for i in range(len(eig_val_sc)):
2 eigv = eig_vec_sc[:,i].reshape(1,3).T
3 np.testing.assert_array_almost_equal(scatter_matrix.dot(eigv), eig_val_sc[i] * eigv,decimal=6, err_msg='', verbose=True)
```
* 得出结果未返回异常,证明计算正确
* 注:np.testing.assert_array_almost_equal计算得出的结果不一样会返回一下结果:
```
>>> np.testing.assert_array_almost_equal([1.0,2.33333,np.nan],
... [1.0,2.33339,np.nan], decimal=5)
...
<type 'exceptions.AssertionError'>:
AssertionError:
Arrays are not almost equal
(mismatch 50.0%)
x: array([ 1. , 2.33333, NaN])
y: array([ 1. , 2.33339, NaN])
```
* 可视化特征向量
```
1 from matplotlib import pyplot as plt
2 from mpl_toolkits.mplot3d import Axes3D
3 from mpl_toolkits.mplot3d import proj3d
4 from matplotlib.patches import FancyArrowPatch
5
6
7 class Arrow3D(FancyArrowPatch):
8 def __init__(self, xs, ys, zs, *args, **kwargs):
9 FancyArrowPatch.__init__(self, (0,0), (0,0), *args, **kwargs)
10 self._verts3d = xs, ys, zs
11
12 def draw(self, renderer):
13 xs3d, ys3d, zs3d = self._verts3d
14 xs, ys, zs = proj3d.proj_transform(xs3d, ys3d, zs3d, renderer.M)
15 self.set_positions((xs[0],ys[0]),(xs[1],ys[1]))
16 FancyArrowPatch.draw(self, renderer)
17
18 fig = plt.figure(figsize=(7,7))
19 ax = fig.add_subplot(111, projection='3d')
20
21 ax.plot(all_samples[0,:], all_samples[1,:], all_samples[2,:], 'o', markersize=8, color='green', alpha=0.2)
22 ax.plot([mean_x], [mean_y], [mean_z], 'o', markersize=10, color='red', alpha=0.5)
23 for v in eig_vec_sc.T:
24 a = Arrow3D([mean_x, v[0]], [mean_y, v[1]], [mean_z, v[2]], mutation_scale=20, lw=3, arrowstyle="-|>", color="r")
25 ax.add_artist(a)
26 ax.set_xlabel('x_values')
27 ax.set_ylabel('y_values')
28 ax.set_zlabel('z_values')
29
30 plt.title('Eigenvectors')
31
32 plt.show()
```
* 结果:
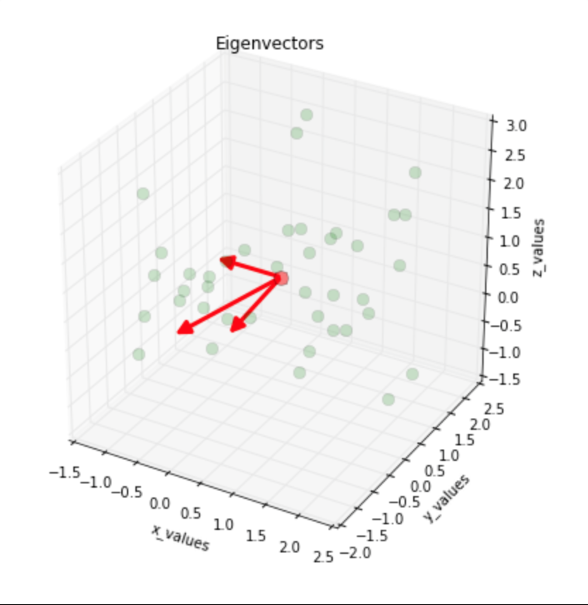
### 7.根据特征值对特征向量降序排列
* 我们的目标是减少特征空间的维度,即通过PCA方法将特征空间投影到一个小一点的子空间里,其中特征向量将会构成新的特征空间的轴。然而,特征向量只会决定轴的方向,他们的单位长度都为1,可以用代码检验一下:
```
1 for ev in eig_vec_sc:
2 numpy.testing.assert_array_almost_equal(1.0, np.linalg.norm(ev))
```
* 因此,对于低维的子空间来说,决定丢掉哪个特征向量,就必须参考特征向量相应的特征值。通俗来说,如果一个特征向量的特征值特别小,那它所包含的数据分布的信息也很少,那么这个特征向量就可以忽略不计了。常用的方法是根据特征值对特征向量进行降序排列,选出前k个特征向量。
```
# 生成(特征向量,特征值)元祖
eig_pairs = [(np.abs(eig_val_sc[i]), eig_vec_sc[:,i]) for i in range(len(eig_val_sc))]
#对(特征向量,特征值)元祖按照降序排列
eig_pairs.sort(key=lambda x: x[0], reverse=True)
#输出值
for i in eig_pairs:
print(i[0])
```
* 结果:
```
1 84.5729942896
2 39.811391232
3 21.2275760682
```
### 8.选出前k个特征值最大的特征向量
* 本文的例子是想把三维的空间降维成二维空间,现在我们把前两个最大特征值的特征向量组合起来,生成d\*k维的特征向量矩阵W
```
1 matrix_w = np.hstack((eig_pairs[0][1].reshape(3,1), eig_pairs[1][1].reshape(3,1)))
2 print('Matrix W:\n', matrix_w)
```
* 结果:
```
1 >>> print('Matrix W:\n', matrix_w)
2 Matrix W:,
3 array([[-0.62497663, 0.2126888 ],
4 [-0.44135959, -0.88989795],
5 [-0.643899 , 0.40354071]]))
```
### 9.将样本转化为新的特征空间
* 最后一步,把2\*3维的特征向量矩阵W带到公式中,将样本数据转化为新的特征空间
```
1 matrix_w = np.hstack((eig_pairs[0][1].reshape(3,1), eig_pairs[1][1].reshape(3,1)))
2 print('Matrix W:\n', matrix_w)
3
4
5 transformed = matrix_w.T.dot(all_samples)
6 assert transformed.shape == (2,40), "The matrix is not 2x40 dimensional."
7
8
9 plt.plot(transformed[0,0:20], transformed[1,0:20], 'o', markersize=7, color='blue', alpha=0.5, label='class1')
10 plt.plot(transformed[0,20:40], transformed[1,20:40], '^', markersize=7, color='red', alpha=0.5, label='class2')
11 plt.xlim([-4,4])
12 plt.ylim([-4,4])
13 plt.xlabel('x_values')
14 plt.ylabel('y_values')
15 plt.legend()
16 plt.title('Transformed samples with class labels')
17
18 plt.show()
```
* 结果:
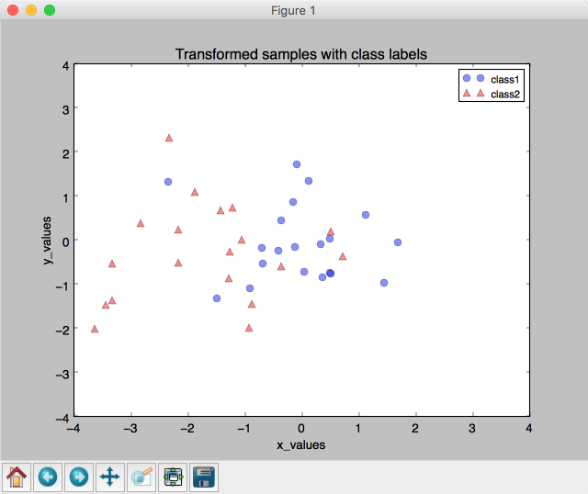
* 到这一步,PCA的过程就结束了。其实python里有已经写好的模块,可以直接拿来用,但是我觉得不管什么模块,都要懂得它的原理是什么。matplotlib有[matplotlib.mlab.PCA()](http://www.cnblogs.com/https:/www.clear.rice.edu/comp130/12spring/pca/pca_docs.shtml),sklearn也有专门一个模块[Dimensionality reduction](http://scikit-learn.org/stable/modules/decomposition.html#decompositions)专门讲PCA,包括传统的PCA,也就是我上文写的,以及增量PCA,核PCA等等,除了PCA以外,还有ZCA白化等等,在图像处理中也经常会用到。
* 最后推荐一个博客,动态展示了PCA的过程:http://setosa.io/ev/principal-component-analysis/ 写的也很清楚,可以看一下;再推荐一个维基百科的,
https://en.wikipedia.org/wiki/Principal\_component\_analysis
